Forschung
Der Zellkern wird von einer Kernhülle umgeben, die das Zytoplasma vom Inneren des Zellkerns trennt. Diese Trennung erfordert effiziente und selektive Transportschleusen, die die Orte der Gentranskription und Proteintranslation miteinander verbinden. Kernporenkomplexe erfüllen diese Funktion, indem sie den Transport von Proteinen und Nukleinsäuren ermöglichen und gleichzeitig inerte Moleküle von der Passage ausschließen. Zudem sind Kernporenproteine an der Genregulation beteiligt, was erklärt, warum viele von ihnen in die Pathogenese bösartiger Tumore sowie anderer menschlicher Erkrankungen wie Vorhofflimmern, vorzeitiger Alterung und Muskel- oder Lipodystrophien involviert sind. Oft sind die zugrunde liegenden molekularen Krankheitsmechanismen noch nicht vollständig verstanden.
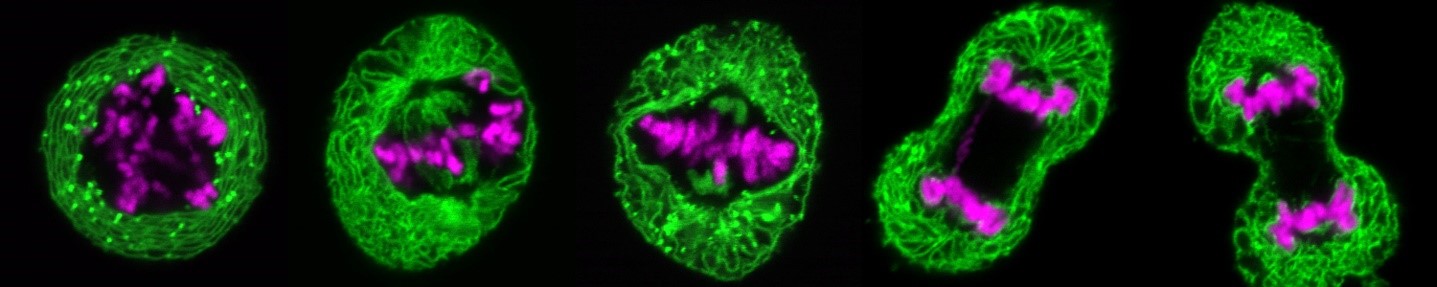
Während der Zellteilung erfahren der Zellkern menschlicher und tierischer Zellen bemerkenswerte morphologische und funktionelle Veränderungen: Zu Beginn der Mitose zerfallen die Kernporenkomplexe sowie die Kernhülle, während das Chromatin kondensiert, um die Bildung der mitotischen Spindel zu ermöglichen, welche die DNA auf die beiden entstehenden Tochterzellen verteilt. Am Ende der Mitose muss die Interphasenstruktur des Zellkerns wiederhergestellt werden, um die Fähigkeit zur DNA-Replikation und -Transkription zu gewährleisten. Dies umfasst unter anderem die Dekondensation des stark verdichteten mitotischen Chromatins, die Neubildung einer geschlossenen Kernhülle um das dekondensierende Chromatin sowie den gleichzeitigen Wiederaufbau der Kernporenkomplexe und deren Integration in die sich neu formierende Kernhülle. All dies geschieht parallel und scheinbar gut koordiniert innerhalb eines kurzen Zeitfensters im Zellzyklus. Viele beteiligte Faktoren sowie deren molekulare Mechanismen – insbesondere hinsichtlich Regulierung und Koordination – sind bestenfalls rudimentär verstanden.
Wir untersuchen verschiedene Aspekte des Wiederaufbaus des Zellkerns, um diese Prozesse in ihrer zellulären Komplexität zu verstehen, molekulare Mechanismen zu entschlüsseln und ein detailliertes Bild der relevanten Faktoren sowie deren Interaktionen zu generieren. Hierfür setzen wir eine Vielzahl biochemischer (z.B. Protein-Protein-Interaktionsassays, Membranbindungsassays, Enzymkinetik) sowie zellbiologischer Experimente (z.B. Live-Cell-Imaging, Zelltransfektionen sowie Knock-outs und Knock-downs) ein. Dabei schließen wir insbesondere die methodische Lücke zwischen gut definierten minimalistischen biochemischen Experimenten und sehr komplexen Untersuchungen in lebenden Zellen. Neben Säugetierzellen nutzen wir auch frisch abgelaichte Froscheier zur Gewinnung von Zellextrakten für verschiedene zellfreie Rekonstitutionsexperimente. In diesen Experimenten lassen sich hochkomplexe zelluläre Prozesse wie Chromatindekondensierung oder der Wiederaufbau der Kernhülle am Ende der Mitose maßgeschneidert im Reagenzglas nachstellen und ihre molekularen Mechanismen analysieren.
Kernporenkomplexe
Kernporenkomplexe zählen mit einer Größe von bis zu 125 MDa zu den größten Proteinkomplexen in Zellen. Ihr schrittweiser und koordinierter Zusammenbau aus über fünfhundert Einzelmolekülen sowie deren Integration in die Kernhülle sind beeindruckende Beispiele für molekulare Selbstorganisation. Wir analysieren diesen Prozess mithilfe biochemischer und zellbiologischer Methoden, darunter zellfreie Assays und Live-Cell-Imaging. Dabei definieren wir die Rolle der Transmembranproteine innerhalb der Kernporenkomplexe und untersuchen, wie Kernporenproteine häufig über amphipathische Helices mit den Membranen der Kernhülle interagieren. Diese Interaktion ist entscheidend für die starke Krümmung der Kernmembranen zu einer Porenstruktur.
Während viele Kernporenproteine eine Diffusionsbarriere im Zentrum der Kernpore bilden, um den Transport der meisten Proteine zu verhindern, ermöglichen einige wenige den Transport, indem sie einen Aktivitätsgradienten der kleinen GTPase Ran an der Kernhülle aufbauen. Wir erforschen, wie das Kernporenprotein Nup50 den Kerntransport stimuliert, indem es auf der Zellkernseite der Pore Ran aktiviert, bindet und für den Transportvorgang zur Verfügung stellt.
Einige Mutationen in Nukleoporinen sind mit erblichen Erkrankungen assoziiert. Wir untersuchen beispielsweise, wie solche Mutationen zum nephrotischen Syndrom bei Kindern führen oder Amyotrophe Lateralsklerose verursachen können. Dabei analysieren wir, ob sie Defekte im Zusammenbau von Kernporen oder Fehlfunktionen in nukleären Transportwegen hervorrufen und warum diese Mutationen in Nieren- beziehungsweise Nervenzellen besonders gravierende Auswirkungen haben.

Weiterführende Literatur:
Kutay U, Jühlen R, Antonin W. (2021). Mitotic disassembly and reassembly of nuclear pore complexes. Trends in Cell Biology S0962-8924(21)00139-2. doi: 10.1016/j.tcb.2021.06.011
Hamed M & Antonin W. (2021). Dunking into the Lipid Bilayer: How Direct Membrane Binding of Nucleoporins Can Contribute to Nuclear Pore Complex Structure and Assembly. Cells. 10(12):3601. doi: 10.3390/cells10123601.
Holzer G, De Magistris P, Gramminger C, Sachdev R, Magalska A, Schooley A, Scheufen A, Lennartz B, Tatarek-Nossol M, Lue H, Linder MI, Kutay U, Preisinger C, Moreno-Andrés D, Antonin W. (2021). The nucleoporin Nup50 activates the Ran guanine nucleotide exchange factor RCC1 to promote NPC assembly at the end of mitosis. EMBO Journal e108788. doi: 10.15252/embj.2021108788
Braun DA, Sadowski CE, Kohl S, Lovric S, Astrinidis SA, Pabst WL, Gee HY, Ashraf S, Lawson JA, Shril S, Airik M, Tan W, Schapiro D, Rao J, Choi WI, Hermle T, Kemper MJ, Pohl M, Ozaltin F, Konrad M, Bogdanovic R, Büscher R, Helmchen U, Serdaroglu E, Lifton RP, Antonin W, Hildebrandt F (2016). Mutations in nuclear pore genes NUP93, NUP205 and XPO5 cause steroid-resistant nephrotic syndrome. Nature Genetics, 48 (4): 457-465.
Kernhüllendynamik
Zu Beginn der Mitose zerfällt die Kernhülle, sodass ihre Protein- und Lipidbestandteile ins endoplasmatische Retikulum aufgenommen werden. Am Ende der Mitose bildet sich in jeder entstehenden Tochterzelle eine neue Kernhülle aus dem endoplasmatischen Retikulum, die das Chromatin umschließt. Wir untersuchen, welche Proteine der Kernhülle und welche Veränderungen am Chromatin erforderlich sind, um die Kernhülle aufzulösen und wieder aufzubauen. Dabei setzen wir insbesondere hochauflösendes Live-Cell-Imaging in Kombination mit quantitativer, automatisierter Bildanalyse ein, um dynamische zelluläre Prozesse zeitlich und räumlich präzise zu erfassen. Zu diesem Zweck entwickeln wir Open-Source-Softwaretools, die auf Deep Learning basieren. Diese Tools ermöglichen es uns, die verschiedenen Phasen der Mitose automatisiert zu erkennen sowie große Datenmengen, die beim Live-Cell-Imaging anfallen, effizient zu analysieren. Dadurch können wir quantitative Parameter extrahieren und anschaulich visualisieren. Auf diese Weise sind wir in der Lage, Defekte während der Mitose und in anderen Phasen des Zellzyklus präzise zu identifizieren.
Weiterführende Literatur:
De Magistris P and Antonin W (2018). The Dynamic Nature of the Nuclear Envelope. Current Biology 28(8): R487–R497
Moreno-Andrés D, Bhattacharyya A, Scheufen A, Stegmaier J (2022). LiveCellMiner: A new tool to analyze mitotic progression. PLoS One 7;17(7):e0270923. doi: 10.1371/journal.pone.0270923
Jose A, Roy R, Moreno-Andrés D, Stegmaier J (2024). Automatic detection of cell-cycle stages using recurrent neural networks. PLoS One. 19(3):e0297356. doi: 0.1371/journal.pone.0297356.
Scheufen A & Moreno-Andrés D (2025). Quantitative Live-Cell Imaging to Study Chromatin Segregation and Nuclear Reformation. Methods Mol Biol . 2025:2874:47-60. doi: 10.1007/978-1-0716-4236-8_5
Chromatindynamik in der Mitose
In menschlichen Zellen ist das mitotische Chromatin im Vergleich zur Interphase bis zu fünfzigmal stärker verdichtet. Am Ende der Mitose dekondensiert es, um den Zustand wiederherzustellen, der für Transkription und Replikation erforderlich ist. Obwohl dieser Prozess entscheidend für die Zellteilung ist, sind die daran beteiligten Faktoren weitgehend unbekannt.
Wir untersuchen, welche Faktoren an der Chromatindekondensation beteiligt sind und welche Reaktionen sie vermitteln. Dabei analysieren wir auch, welche Chromatinveränderungen einschließlich Histon- und DNA-Modifikationen während der Dekondensation notwendig sind und wie diese Prozesse in einem funktionalen und regulatorischen Zusammenhang mit der Neubildung der Kernhülle und den Kernporenkomplexen stehen. Da RNA-Protein-Komplexe sowie RNA-Helikasen eine wesentliche Rolle bei der Dekondensierung auf der Oberfläche der mitotischen Chromosomen spielen, konzentrieren wir uns insbesondere auf die Analyse dieser RNA-Protein-Komplexe und darauf, welche RNAs hierbei vorrangig beteiligt sind. Für unsere Untersuchungen setzen wir zellfreie Assays sowie Experimente in Säugetierzellen ein, darunter RNAi- und inhibitorbasierte Screenings sowie Live-Cell-Imaging.

Weiterführende Literatur:
Antonin W and Neumann H (2016). Chromosome condensation and decondensation during mitosis. Current Opinion in Cell Biology, 40:15-22.
Magalska, A, Schellhaus, AK, Moreno-Andres, D, Zanini, F, Schooley, A, Sachdev, R, Schwarz, H, Madlung, J, Gerken, J and Antonin, W (2014). RuvB-like ATPases function in chromatin decondensation at the end of mitosis. Developmental Cell, 31 (3): 305-318.
Jühlen R, Wiesmann S, Scheufen A, Stausberg T, Braun I, Strobel C, Llera-Brandt C, Rappold S, Suluyala R, Tatarek-Nossol M, Lennartz B, Lue H, Schneider M, Perez-Correa F, Moreno-Andres D, and Antonin W (2025). The DEAD-box helicase eIF4A1/2 acts as RNA chaperone during mitotic exit enabling chromatin decondensation. Nat Comm doi: 10.1038/s41467-025-57592-1.



